2024/10/26(土);初稿
2026/01/06(火);タイトル変更、サムネイル変更
はじめに
TCGA(The Cancer Genome Atlas Program)は公的データベースであり、誰でも利用可能である。トランスレーショナルスタディーや、それに限らず基礎研究に対してもこんなに有用なものはない。オープンアクセスのデータだけでなく、限られたユーザーがアクセスできるデータ(Controlled data;オープンアクセスではないデータ)もあるが、それを使わなくても十分な解析ができると考えている。自分のような医療系につま先を突っ込んだ人間にとっては、もはや宝の山に見える。
以下に、TCGAからのRNAシークエンスのカウントデータをウンロードする方法を記す。TCGAに限らず、TARGETやCPTACのデータも同様にダウンロードできる。もちろん、RNAシークエンスではなく変異解析のデータなども同様にダウンロード可能である。自分はRNAシークエンスを主に扱っているので、それについて述べる。
SWAP領域の設定
生存率の解析までは一般的なコンピューターでも十分に可能である。一般的なコンピューターとはintel core i5とか、core i7とか、メモリを8GB以上、ハードディスクを512BG程度積んでいるようなノートパソコンである。しかしながら、8GBでは計算中にメモリ不足で止まるのは明らかであり、そこはスワップ領域を設定する(Windowsでもこのような呼び方なのかはよくわからない。)必要がある。こういった大きなデータセットを用いた解析ではスワップ領域を設定しないと解析はほぼ止まるのではないかと思う。自分の経験では、TCGAのデータセットを使ってsingle sample GSEAを使う場合、120GBまでメモリを消費した。その倍くらいは最低でも必要になるだろうと思う。設定方法は別の記事に記したので、必要ならば参考にしたら良いと思う。
Windowsに関する愚痴
Windowsの優秀さを実感したのが、このメモリ管理に関してのスムーズさである。特に、WindowsとCore i9などのようなEコアやPコアが並んでいるCPUとの相性は、Ubuntuなどよりも明らかに良く、それはRを使っていても実感するところである。最近ではRだけを使うならばUbuntuなんか使わずにWindows使えばかなり早いのではないかと本気で思っているくらいである。
なんでWindowsはこんなに優秀なOSなのに、エクスプローラーだの、PowerShellだの、下句OneDriveなどと、あんなに使いにくいものばかりをユーザーに使わせるのだろう。もういっそのこと、Microsoftは解析用Windows OSみたいなのを売り出したら良いのではないだろうか。
Microsoftという大企業も、Me、 Vista、8、11のような世間では駄作と言われているような製品を作り(個人的には11が一番嫌い。VistaはXPとそんなに違わないし、8のデザインについては会社のデシジョンが完全にどうかしていると思わせてくれるところだが、OS自体が超軽いので嫌いじゃない。しかし、全ての駄作に共通しているのが、ユーザーのことを全く考えていないというところである。)、XP、7、10で改善する(XPが一番合理的だったと思う)ことで個人や部署の業績にするという、しょうもない業績管理システムを採用してるんだろうな、と思うところである。これは、わざと改悪してそれを改善することで業績を得るという、日本の大学や企業の人事や総務でも常態的に行われているだろう仕事の仕方である。さしずめ、次のWindows OSは非常に良い出来になるはず。少なくともOne DriveとかSurfaceという****がなんとかなるだろう。
マニフェストファイル(manifest file)とそれに紐づく検体や臨床情報(clinical.tsv、sample.tsv、followup.tsvなど)をダウンロードする。
まずは、TCGAから解析に必要なデータをダウンロードする必要がある。まず、最も重要と考えられるファイルが、manifest file(マニフェストファイル)である。これには、ダウンロードするカウントデータの情報が記載されており、これを後述のgdc-clinetに渡すことで、必要なデータを一気にダウンロードすることができる。個人的に思うのが、間違ってもChomeやSafariなどのWebブラウザから一つ一つを個別にダウンロードするなんてことは、当然ながら避けなければならない。その理由は、個別に選んでいたら、一生に近い時間がかかってしまうためである。ダウンロード自体も、ウェブブラウザから選んでダウンロードする場合、コマンドラインからダウンロードした場合に比べて時間もかかるような気もするし、gdc-clientを使えば、ダウンロードのログを取ることもできるためである。データが大量になり、どこかでエラーが出ているかもしれないので、このログは重要である。
TCGA
この記事を書いている時点でのTCGAのバージョンは、v41.0である。これらは論文にまとめる際に重要になってくるはずなので、ダウンロードした日は忘れずにMarkdownか実験ノートにかいておくべきである。バージョンの履歴はここに書いてある。https://docs.gdc.cancer.gov/Data/Release_Notes/Data_Release_Notes/
まずはTCGAにアクセスし、「Access TCGA Data」を押す。
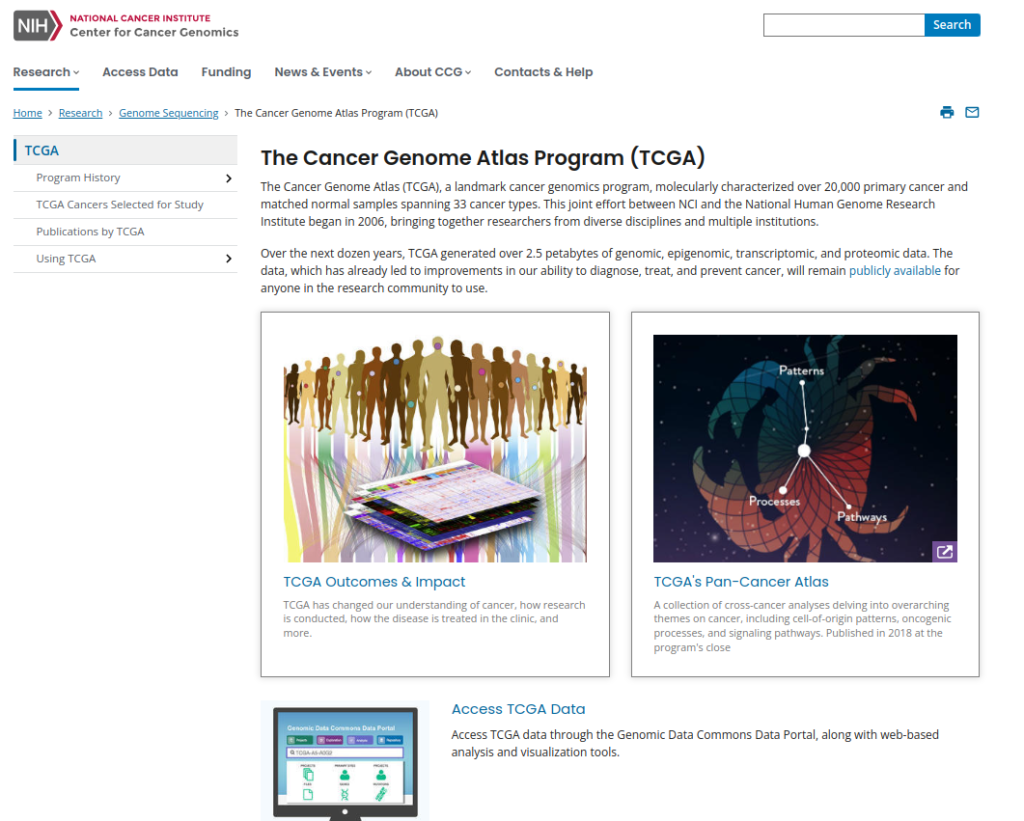
そうすると以下のような画面になるはずなので、Cohort Builderを押す。
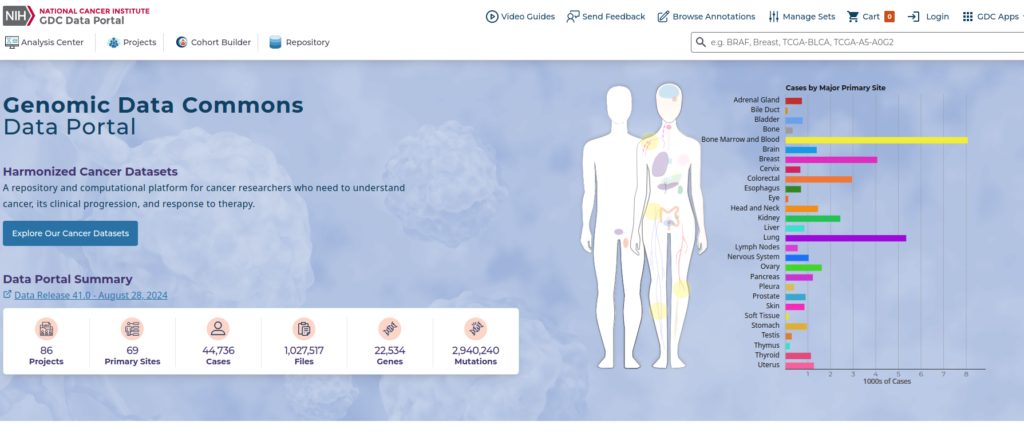
Cohort Builder
ここのProgramからTCGAを選択し、フロッピーディスクのボタンで選択したProgramを保存する。ここには、20241019TCGAという名前で選んだProgramを保存した。

Repository
次にRepositoryに行き、左側に配置されてている「Experimental Strategy」などから、目的の項目を選ぶ。
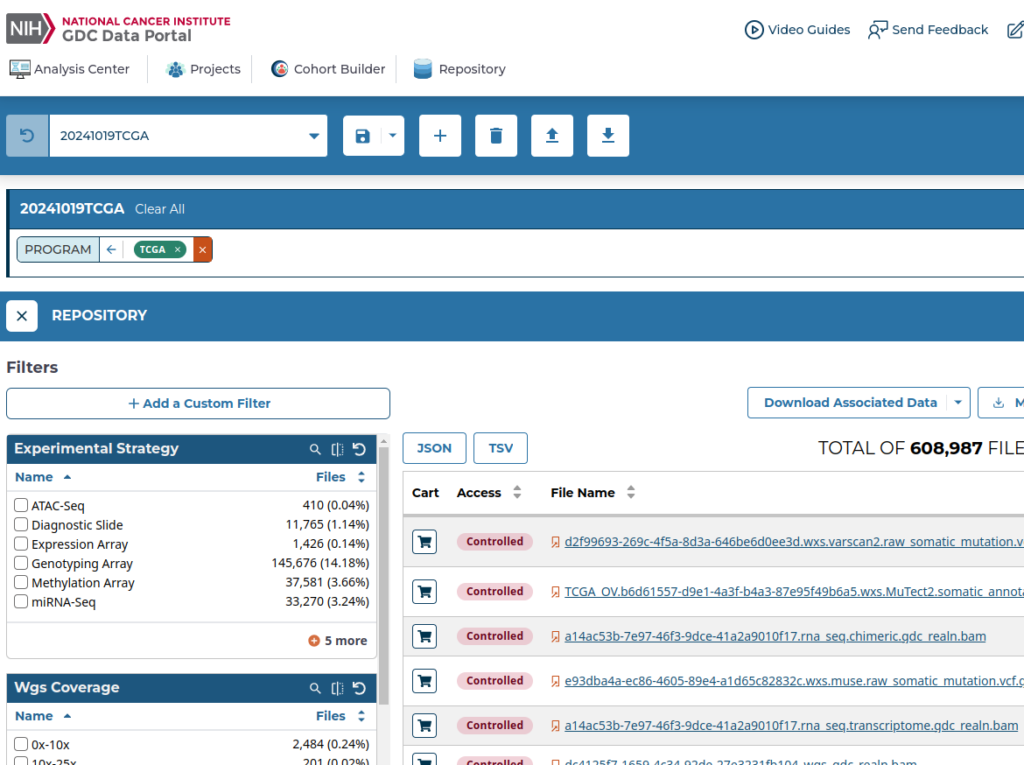
各疾患における遺伝子発現量に関わる解析を行うためには、以下に記すような項目を選べば良いように思う。当然、他の目的がある場合はその目的に応じた項目を選ぶ必要がある。
Preservation Methodが2つあるのは、症例(Case)を全部一度にCartに保存しきれないためである。一度に保存できる症例は10000例であり、全症例を選ぶとその数を超えてしまう。なので、ここでは2分割してCartにい入れることにする。このManifestファイルが分割されていることはそんなに問題ではない。Manifestファイルが分割されていてもデータは個々のディレクトリでダウンロードされてくるので、ダウンロードしたファイルを保存するディレクトリを同じ場所に指定すれば良いだけである。
Group 1
| 項目 | 値 | その他 |
|---|---|---|
| Experimental Strategy | RNA-seq | |
| Data Category | sequenceing reads transcriptome profiling | |
| Data format | tsv | |
| Workflow type | STAR – counts | |
| Access | open | |
| Preservation Method | ffpe oct | Group 1 |
Group 2
| 項目 | 値 | その他 |
|---|---|---|
| Experimental Strategy | RNA-seq | |
| Data Category | sequenceing reads transcriptome profiling | |
| Data format | tsv | |
| Workflow type | STAR – counts | |
| Access | open | |
| Preservation Method | unknown | Group 2 |
症例を選んだら「Add All Files to Cart」を押してCartに入れ、そのCartに移動する。
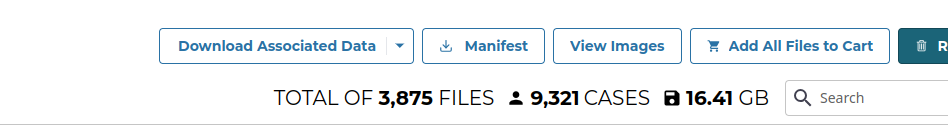
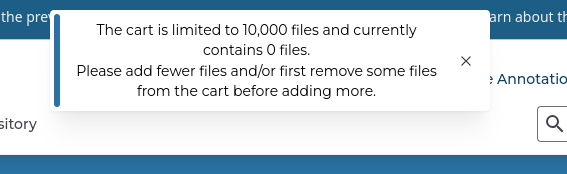
カートに移動し、Download Associated DataからClinical: TSV、Biospeciemen: TSV、Sample Sheet Metadataを、そして、Download CartからManifestをダウンロードする。分割1のこれらのデータのダウンロードが終わったら、Remove From Cartでカートの内容を全部消し、次にRepositoryに戻って分割2のグループを選び、それらをカートにいれ、同じようにClinical:TSV、Biospeciemen:TSV、Manifestなどをダウンロードする。
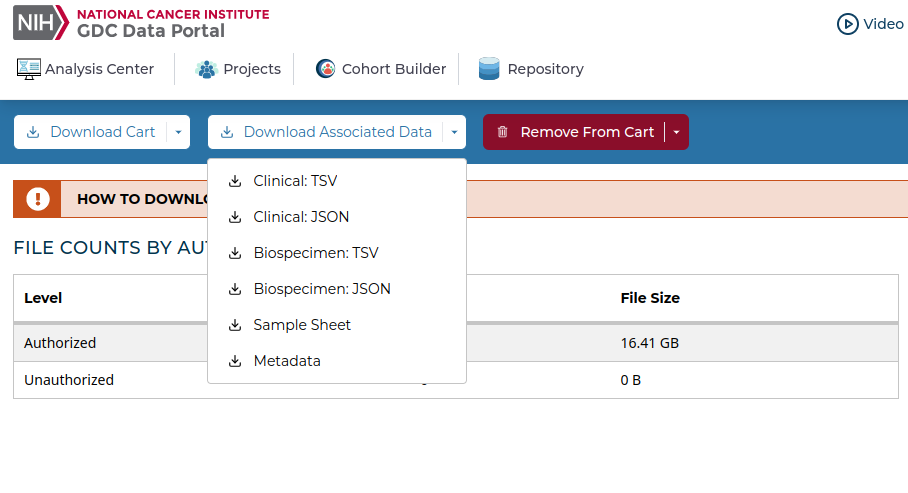
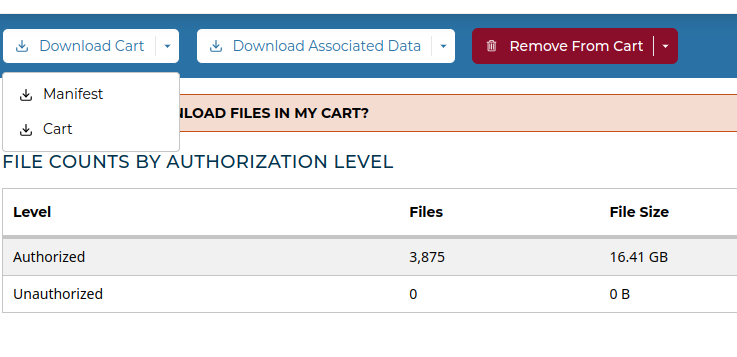
gdc-clientをインストールしデータをダウンロードする。
ダウンロードではGDC-clientというソフトを使用する。Windowsの場合、実行ファイルがダウンロードできるので、それを使用する。正直、これはLinuxの方がスムーズに使えるように思うので、WSL(Windows Subsystem for Lunux)を入れている場合は100%WSLのほうが便利である。とはいえ、例えば、職場や大学でWSLを自由にインストールできない、という人も多いのではないかと思う。なので、一番何度が高いWindowsでのgdc-clientの使用方法をここで述べておく。Macは、Linuxとほぼ同じである。
自分は主にLINUX(Ubuntu 22.04)を中心に、しゃあなしでWindowsを使うといった具合であり、Ubuntu 22.04が第一優先である。そのため、この記事もUbuntu 22.04を主に使用する前提で話を進めていく。WindowsとMacについてはインストールと使い方のみを述べる。MacについてはUbuntuと同じbashを使うためほぼ同じと理解して良い。WindowsとMacが不要ならば、それは無視してOKである。
Windows (Windows 10)
最初にWindowsを述べる。初っ端からちょっと特殊である。
インストール
ここ(https://gdc.cancer.gov/access-data/gdc-data-transfer-tool)からバイナリーをダウンロードする。現状では、Windows x64のところにあるバイナリをダウンロードする。
インストールは、圧縮してあるファイルを展開し、実行ファイル(gdc-client.exe)を任意のディレクトリにコピーするだけである。
UbuntuやMacなどに慣れていると、ここからが若干の落とし穴な気がする。UbuntuやMacでは、そのバイナリを保存しているディレクトリにパスを通して…という作業をする。Windowsでも環境変数からパスを通すことができるのだが、このgdc-client.exeのパスを通しても、その後ログオフや再起動までしても一向にgdc-client.exeで起動することができない。なので、作業するディレクトリに直接gdc-client.exeを保存し、そこにchdirで移動し、そこでダウンロードの作業を行う、ということをしなければならなかった。
自分はユーザー直下にGDCというディレクトリ(フォルダ)を作成し、そこにgdc-client.exeを保存し、gdc-client.exeを使う場合はそのGDCというディレクトリにchdirしてから実行した。これで上手く行った。
使い方
chdir C:\Users\Someone\GDC # At first, change current directory to GDC directory.
gdc-client.exe download -m manifest.txt --log-file LOG_FILE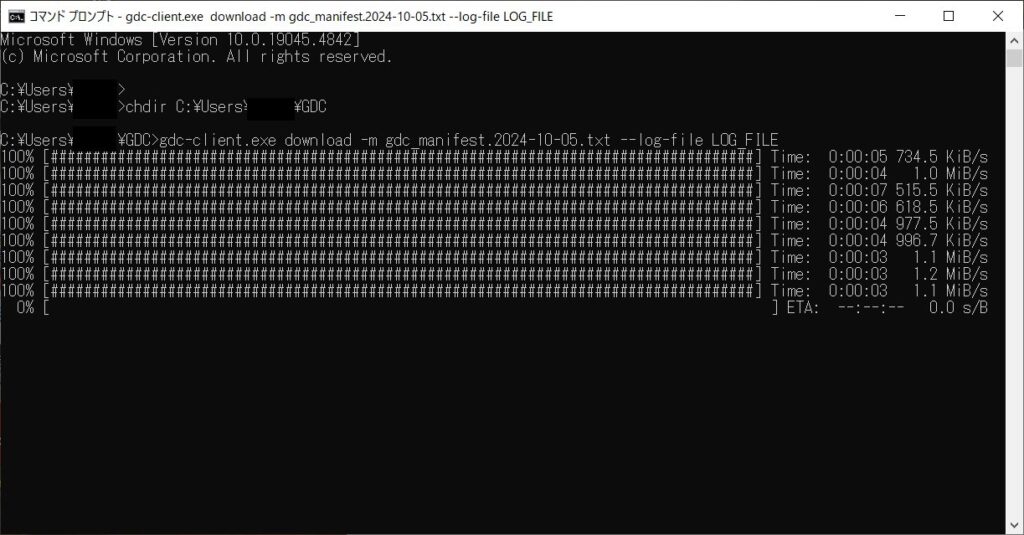
Mac (MacOS Monterey)
まず、これはMacOS Monterey(12.7.6)なので、最新のOSとはどこか違う可能性があるので注意する必要がある。自分はMacはまだこれを使っている。もう古くなってきたと思う。
インストール
ここ(https://gdc.cancer.gov/access-data/gdc-data-transfer-tool)からバイナリーをダウンロードする。
IntelのCPUとApple M2とかM3だとかのApple Siliconを使っている場合とでバイナリーを分ける必要がありそう。もうぼちぼちIntel CPUが少数派になってくるのだろう。というか、Apple M2とかに対応したライブラリってどのくらいあるのだろうか。Ubuntuとかと同等に動くのだろうか。結構気になるところである。まぁ、言うても、今とのところMacを本気で解析に使う予定はないのであまり気にはしていないが。それに、シングルセルRNAシークエンスにしてもこのTCGAにしても、どんどん扱うデータが大きくなり、ライブラリも高度になり、ノートPCレベルでは解析が追いつかなくなるに決まってる。そうなれば、おそらく今後はUbuntu一択になっていくと思う。
インストール自体は、ダウンロードしたファイルを展開し、バイナリーを任意のディレクトリに保存し、そのパスを通せばOKである。ここでは、自分のユーザーディレクトリの直下にGDCというディレクトリを新しく作り、そこにgdc-clientを保存した。パスは以下のように通す。ただし、何故かこれもうまく動かなかった。
cd # Move to current directory
open -a TextEdit .bash_profile # Open ,.bash_prifile file using TextEdit
# Add PATH=$PATH:/Users/Someone/GDC/gdc-client, and save.
# "Someone" is probably your user directory.使い方
Pathを通したにも関わらず、シンプルにgdc-clientと打っても何故かプログラムが走らない。そんなときは、バイナリを保存したディレクトリに直接行き、./gdc-clientと打てば良いと経験上知っている。ということで、それを行ってみると、はやり実行することができた。
cd /Users/Someone/GDC/ # or cd GDC
./gdc-client download -m "/Volumes/Someone/Blog/TCGA/gdc_manifest.txt" -d "/Users/Someone/GDC/" --log-file LOG_FILEMacの場合、ここで重要な注意点がある。初めてgdc-clientを実行する場合、絶対にセキュリティーのエラーが出る。なので、「システム環境設定」、「セキュリティとプライバシー」、「ダウンロードしたアプリケーションの実行許可:」、「App Storeと確認済みの開発元からのアプリケーションを許可」にチェックを入れる必要がある。それで、改めて上記のコードを流すと、また実行してよいかどうか効かれるので、それで許可を与えれば良い。そうすれば次回からいちいち聞いてこなくなる。
Ubuntu 22.04
インストール
ここ(https://gdc.cancer.gov/access-data/gdc-data-transfer-tool)からバイナリーをダウンロードし、どこか都合の良いディレクトリに入れる。homeにGDC_Clientというディレクトリを作っても良いし、単純にhomeの中でも良いように思う。動けばOK。バイナリをhomeのどこかに保存し、次にパスを通す。
パスはhome直下にある.bashrcをgedit(何でもOK)などで開き、PATH=$PATH:/home/path/gdc-clientのように、バイナリを保存した場所を記載して、geditを保存すればよい。いつも忘れるので、一応以下に書いておく。
cd
sudo gedit .bashrc # open .bashrc using gedit
# add PATH=$PATH:/home/path/gdc-client into .bashrc, and save it.
echo $PATH # check registered PATH
source .bashrc # be effective the path.
使い方
以下を流す。/mnt/path/to/TCGA_data/というディレクトリに1、2、countというディレクトリを作っておく。1にはダウンロードしたGroup 1のマニフェストファイル、2にはダウンロードしたGroup 2のマニフェストファイルが入っている。countというディレクトリはダウンロードされてくるファイルが入る。logファイルは保存したほうが良い。かなりの数のファイルをダウンロードすることになるので、エラーが全く無いほうが珍しい。後述するが、Group 2のダウンロードのときに64個のファイルがダウンロードに失敗していた。どのファイルがダウンロードに失敗したかを見つけるためには、このlogファイルを見る必要がある。
# Download Group 1
gdc-client download -m "/mnt/path/to/TCGA_data/1/gdc_manifest.2024-10-19.txt" -d "/mnt/path/to/TCGA_data/count" --log-file "/mnt/path/to/TCGA_data/1/gdc_manifest_1_log.txt"
# Download Group 2
gdc-client download -m "/mnt/path/to/TCGA_data/2/gdc_manifest.2024-10-19.txt" -d "/mnt/path/to/TCGA_data/count" --log-file "/mnt/path/to/TCGA_data/2/gdc_manifest_2_log.txt"そうすると順次ダウンロードが始まる。
上手く行ったらSuccessfully downloadedになる。
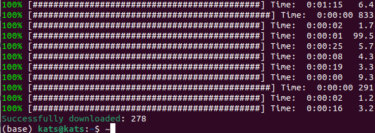
ダウンロードに失敗したファイルがあった場合、このような感じになる。
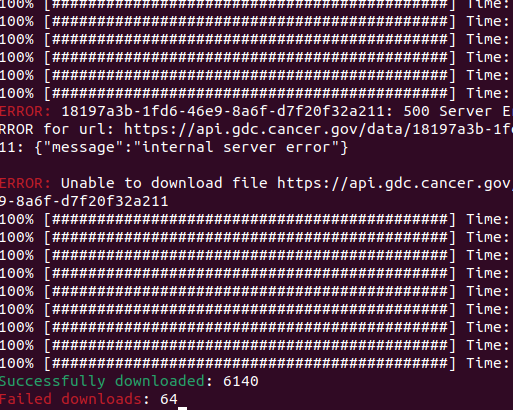
この場合、この64個のファイルをもう一度ダウンロードしなければならない。そんなときに役に立つのが、ログファイルである。これはGroup 2のダウンロードに失敗したファイルなので、gdc_manifest_2_log.txtにその情報が記載されている。
Logファイルからダウンロードに失敗したファイルを抽出し改めてダウンロードする。
まず、Logファイルを眺めてみる。
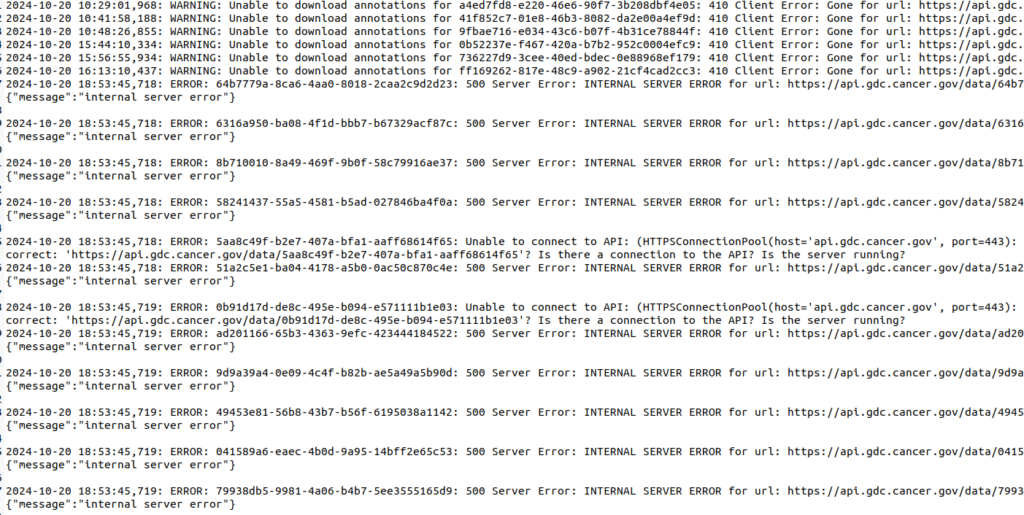
ダウンロードできなかったファイル。
最悪である。こんなものを手作業で選ぶとか唯の地獄である。ということで、以下のRコードを使用した。
library(tidyverse)
# 1
error1 <- read_delim(file = "/mnt/path/to/TCGA_data/1/gdc_manifest_1_log.txt", delim = ": ", col_names = FALSE)
manifest1 <- read_tsv(file = "/mnt/path/to/TCGA_data/1/gdc_manifest.2024-10-19.txt")
manifest1_retry <- manifest1[manifest1$id %in% error1$X3,]
# 2
# first 6 line is warning. so skip 6 line at the beginning.
error2 <- read_delim(file = "/mnt/path/to/TCGA_data/2/gdc_manifest_2_log.txt", skip = 6, delim = ": ", col_names = FALSE)
manifest2 <- read_tsv(file = "/mnt/path/to/TCGA_data/2/gdc_manifest.2024-10-19.txt")
manifest2_retry <- manifest2[manifest2$id %in% error2$X3,]
error2_warning <- read_delim(file = "/mnt/path/to/TCGA_data/2/gdc_manifest_2_log.txt", delim = ": ", col_names = FALSE)[1:6,]
error2_warning$X3 <- gsub("Unable to download annotations for ", "", error2_warning$X3)
manifest2_warning_retry <- manifest2[manifest2$id %in% error2_warning$X3,]
manifest2_retry <- rbind(manifest2_retry, manifest2_warning_retry) %>% distinct(id, .keep_all = TRUE)
manifest_retry <-rbind(manifest1_retry, manifest2_retry)
dir.create("/mnt/path/to/TCGA_data/retry")
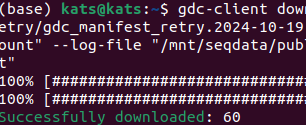
write_tsv(manifest_retry, "/mnt/path/to/TCGA_data/retry/gdc_manifest_retry.2024-10-19.txt")これで、/mnt/path/to/TCGA_data/retry/というディレクトリにgdc_manifest_retry.2024-10-19.txtというManifestファイルが出来上がるので、それを改めてgdc-clientに流す。そうするとダウンロードが上手く行った。実際は60個だったらしい。なんか良く覚えていない。重複があったりしたのだろう。
まとめ
以上がTCGAのRNA-seqのデータのダウンロード方法である。TCGAはRNA-seqだけでなく、DNA-seqの結果(変異の解析結果)も保管されている。これらは基礎研究で得られて結果のトランスレーションにとって、非常に貴重かつ有用であるのは明らかである。これを駆使することができれば、それはそれは強い味方になるはずである。
ここではダウンロードの方法を書いたが、いずれは解析自体についても書こうと思う。