2025/10/04(土);初稿
2025/11/08(土);追記;並列化が元から組み込まれているライブラリに対してforeachを使うのはそもそも良くない。
はじめに
以前TCGAやCPTACのデータをダウンロードしたが、最近はこれまで解析を行ってきた解析手順や手法をブログに記録しておくためそのデータを使って色々と解析を行っている。今後のためにと思って、多少無茶な解析でも行っているわけだが、そのときに自分の能力では追いつかないほどのどうしようもない問題に当たってしまった。現在はRyzen 9 9950Xを使っているのだが、インテルのCPUではなく、AMDのCPU(Ryzen 9 9950X)もしくは現在使用しているマザーボード(ASUS PRIME X870-P CSM WIFI)、で起こり得る問題な気がする。同じようなコードをRstudioで走らせても、Core i9 14900Kでは問題が無かったためだ。なんとか解決し、何やったら改善したような気もしないでもないが、なんとなく、腑に落ちない。
症状
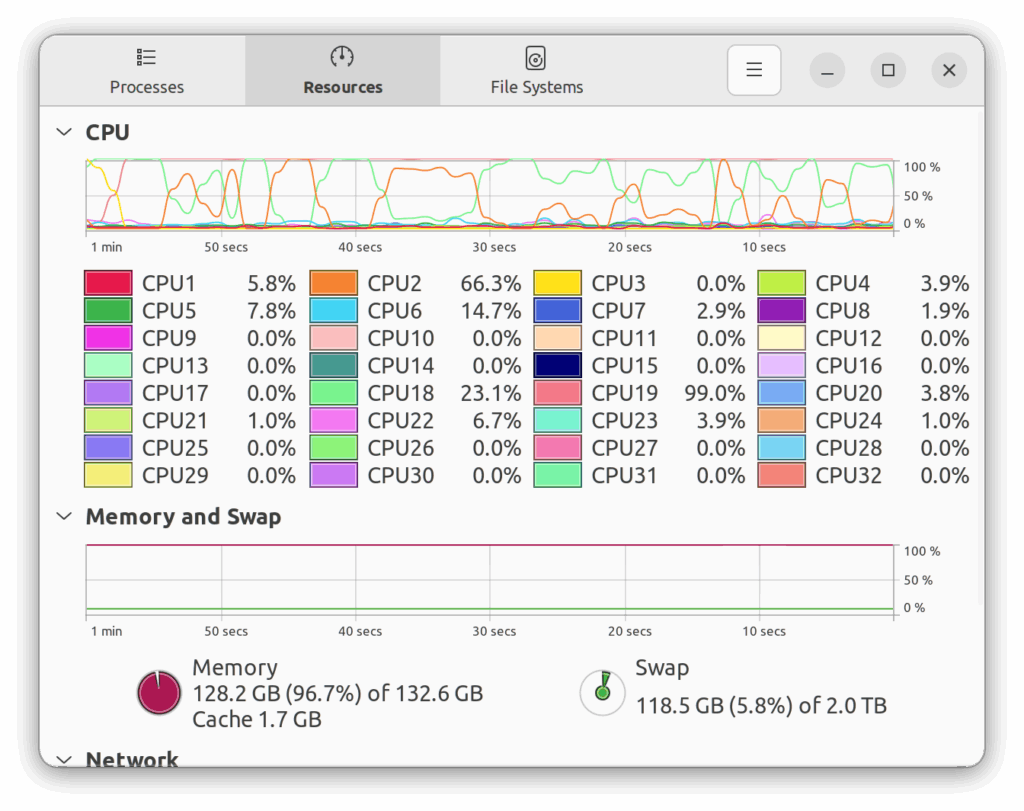
上述のように、これまで行ってきたTCGAの解析方法を記録するために以前ダウンロードしたTCGAのデータをいじっているときに起こった。TCGAからダウンロードして、解析のために整形した10017例の遺伝子発現プロファイルを使い、MsigDBに登録されている遺伝子セットとssGSEA2, escape, fgsea, GSVA, fgseaを用いてGSEAを行っていると、特に、マルチスレッドで計算させようとした場合にRSTUDIOや、時にはコンピューター自体が突然シャットダウンしてしまった。
これを受けて「しゃあないからシングルスレッドでやろうか….」とか色々と考えたが、最終的に「こんな高級コンピューターなのに、シングルスレッドで計算してたら、金も時間ももったいなすぎる…..!!!」と我に返り、Google検索、ChatGPT、BIOSの説明書を調べながら、その原因を探っていくことになった。
マルチスレッドが上手く行っていないことが原因らしい
最近のコンピューターは、既に良く知られているように、マルチコア、マルチスレッドである。マルチコアは、一つのCPUの中に計算するところが複数あるタイプのCPUであり、マルチスレッドとは、各コア毎に複数の計算ユニットを分けて(理論コア、理論プロセッサ)計算するタイプのCPUの形態である。今回起こった症状は、doParallelを使って並列化したときに、一つの理論コアに対してあまりにも多くのタスクを割り当ててしまうこと、すなわち、オーバーサブスクリプションがうまく行っていないことが原因で起こるクラッシュのようだ。さながらインターネットのDoS攻撃みたいなもんだろうか。これを予防するためには、並列化のときに各理論コアに割り当てるタスクを1つに制限する必要がありそうだ。このオーバーサブスクリプションというのは、上手いこと設定されているならば問題ではないらしい。むしろ、CPUを多くのユーザーに割り当てる必要があるサーバーなんかでは、おそらく必須のコントロールであるようだ。
ここまで分かればなんとかなる気がする…!!ということで正味3日くらい費やした結果、上記に加えて、どうやらSMT(Simultaneous MultiThreading)が上手く行っていないかも、という結論に至った。インテルで言うところのハイパースレッディングである。自分のPCはAMDのCPU(Ryzen 9 9950X)なので、ハイパースレッディングではなくSMTで呼ぶらしい。これがわかったときには、良かったと思う反面、自分のCPUをフル活用できていないような気がして、なんか悲しくなった。言うて、計算結果はマルチスレッドのときとあんまり変わらなかったから良いのだが、どこかで何か効率を損なっている気がする…
対処方法
ということで、以下がChatGPTに聞きながら設定したことである。残念ながら、おそらく最適な方法では無いんだろうけど、自分の能力ではこのくらいしか思いつくことが出来なかった。この設定によってパフォーマンスを下げる気がするが、重い計算をシングルスレッドで延々と行うよりはずっと良いと思う。ちょっと残念だけど。
使用したGCTファルとGMTファイル
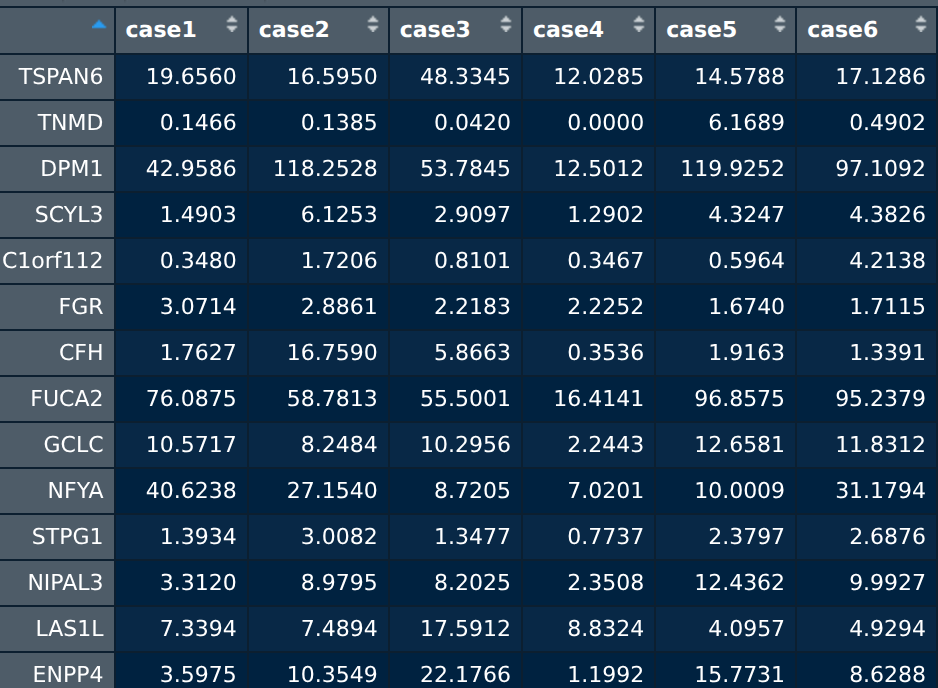
データセットは、TCGAからダウンロードした遺伝子発現プロファイルでる。これを各疾患の生存率に与える影響を解析するために整形したデータである。これは、このときにダウンロードしたデータである(gdc-clientを使ってTCGAの公開データをダウンロードする)。以下のような感じになっている。この遺伝子発現プロファイルを全部結合して、各患者xTPMで正規化された各遺伝子発現量(10017例 x59422遺伝子)のデータフレームもしくはマトリックスである。この作成方法については別にポストしようと思う。ここでは、このデータセットをfgseaとssGSEA2に入れて、ちゃんと並列化して計算できるかどうか調整、テストしようと思う。
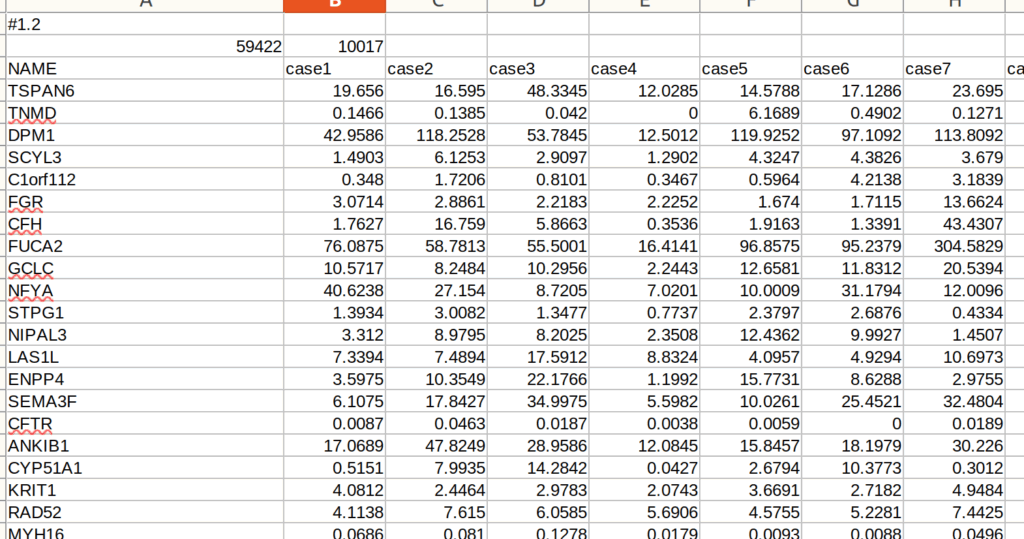
遺伝子シグネチャー(GMTファイルで提供する)としては、MsigDBのHallmark of cancerを使った。これは構成される遺伝子セットの数が50で、C2とかC7とかC8とか他のシグネチャーセットよりも圧倒的に少ないためである。ここ(https://www.gsea-msigdb.org/gsea/msigdb/human/collections.jsp)からダウンロードできる。
doParallelでのCPUのオーバーサブスクリプションを制限する
まずやったことの1つ目は、RでdoParallel()を設定するときに各種並列化ライブラリのマルチスレッドの項目を1にして、オーバーサブスクリプションをしないようにすることである。このテストのためにssGSEA2とfgseaを使用して、動作の確認をするとしよう。ssGEEA2は関数内で並列化が設定されるので、このあたりは自分では設定できない。なので、fgseaを使って試すことにする。以下がそのためのコードである。fesgaの良いところは、対象とするデータセット内の1列ずつGSEAできるところである。つまり、例えばTCGAなどのデータセットなら患者ごと、シングルセルRNAシークエンスのデータセットなら細胞毎にGSEAが可能である。このようにforeach()で全列に対して計算を適用していく。「マトリックスにしてるんだから一気にやれよ面倒くせーなぁ」と言いたくもなるので、悪い点とも言えるが、やっぱり、こっちの最良でforeach()を使えたりするので、やはり利点だろう。ちなみに、これをfor()でやろうとすると、何十倍も計算時間が必要なので、このように10017例 x59422遺伝子もあるデータセットにはfor()はやめたほうが言い。
通常ならば、以下のようにするだろう。これ、以前仕事でCore i9 14900を使っていたときはこれで大丈夫だったんだけど、Ryzen 9 9950XだRstudioもしくはコンピューターがクラッシュして突然落ちた。ちなみに、使っているBIOSとかも一応最新にしてある。
# これは通常版。
## まずはGMTファイルを読み込む。
geneset_fgsea_hallmark <- fgsea::gmtPathways("/mnt/team4tb/Dropbox/Blog/2024 11 02 TCGA analysis/h.all.v2025.1.Hs.symbols.gmt")
## doParallelの設定
core <- getOption("mc.cores",detectCores())
cl <- makeCluster(core, type = "FORK") #FORK #PSOCK
registerDoParallel(cl)
## fgsea
set.seed(1)
result_fgesa_foreach_hallmark <- list()
result_fgesa_foreach_hallmark <- foreach(i = seq_len(ncol(enrich_3_t_mat)), .packages = "fgsea", .inorder = TRUE) %dopar% {
fgsea(pathways = geneset_fgsea_hallmark,
stats = enrich_3_t_mat[, i],
minSize = 10,
maxSize = 500)}
stopCluster(cl)doParallel()でCPUのオーバーサブスクリプションを制限するためには、makeCluster()、registerDoParallel()の次に、それらを以下のように設定すれば良いらしい。ちなみに、これはchatGPTに教えてもらいました。もうバイオインフォマティシャンなんて、ChatGPTで代価出来るから怖い。ほんと、インフォマティクス系の技術員はAIに取って変わられる職業の一つだろうな、と思う。
以下のenrich_3_t_matというデータは、このための整形した10017例 x59422遺伝子のデータセット(マトリックス)である。これにより、fgseaはマルチスレッド全開で流すことができた。
# CPUのオーバーサブスクリプション制限版。
## まずはGMTファイルを読み込む。
geneset_fgsea_hallmark <- fgsea::gmtPathways("/mnt/team4tb/Dropbox/Blog/2024 11 02 TCGA analysis/h.all.v2025.1.Hs.symbols.gmt")
## doParallelの設定
core <- getOption("mc.cores",detectCores())
cl <- makeCluster(core, type = "FORK") #FORK #PSOCK
registerDoParallel(cl)
## doParallelの設定
clusterEvalQ(cl, {
Sys.setenv(OMP_NUM_THREADS="1",
OMP_THREAD_LIMIT="1",
OMP_MAX_ACTIVE_LEVELS="1",
OMP_DYNAMIC=FALSE,
MKL_NUM_THREADS="1",
MKL_DYNAMIC=FALSE,
OPENBLAS_NUM_THREADS="1",
GOTO_NUM_THREADS="1" ,
RCPP_PARALLEL_NUM_THREADS="1")})
## fgsea
set.seed(1)
result_fgesa_foreach_hallmark <- list()
result_fgesa_foreach_hallmark <- foreach(i = seq_len(ncol(enrich_3_t_mat)), .packages = "fgsea", .inorder = TRUE) %dopar% {
fgsea(pathways = geneset_fgsea_hallmark,
stats = enrich_3_t_mat[, i],
minSize = 10,
maxSize = 500)}
stopCluster(cl)環境変数を設定することでCPUのオーバーサブスクリプションを制限する
ここで気がついたことがある。上記のSys.setenv()で設定した項目をコンピューターの環境変数に予めセットしておけば、CPUのオーバーサブスクリプションは1になって、clusterEvalQ()をやる必要がないのでは?ということだ。ということでそれをやってみる。これはRStudioではなくbashで以下のように設定する。
まず、su権限で/etc/Rに移動し、geditでRenvironを開く。
su
cd /etc/R
gedit Renviron次に、以下を加えて保存し、geditを閉じる。
# BLAS / OpenMP
OMP_NUM_THREADS=1
OMP_THREAD_LIMIT=1
OMP_MAX_ACTIVE_LEVELS=1
OMP_DYNAMIC=FALSE
# If you use MKL or OpenBLAS
MKL_NUM_THREADS=1
MKL_DYNAMIC=FALSE
OPENBLAS_NUM_THREADS=1
GOTO_NUM_THREADS=1
# RcppParallel / TBB
RCPP_PARALLEL_NUM_THREADS=1次にbashに戻って、exitする。別にこれを入れる必要は無いように思うが、祈りを込めて丁寧に退出するって感じ。
exit次にRstudioで以下を流して、Rの環境変数を確認する。
Sys.getenv()そしていよいよ、fgseaをforeachで回してみる。
# Renvironに各項目を追加した後なら、これが完遂できた。
## まずはGMTファイルを読み込む。
geneset_fgsea_hallmark <- fgsea::gmtPathways("/mnt/team4tb/Dropbox/Blog/2024 11 02 TCGA analysis/h.all.v2025.1.Hs.symbols.gmt")
## doParallelの設定
core <- getOption("mc.cores",detectCores())
cl <- makeCluster(core, type = "FORK") #FORK #PSOCK
registerDoParallel(cl)
set.seed(1)
result_fgesa_foreach_hallmark <- list()
result_fgesa_foreach_hallmark <- foreach(i = seq_len(ncol(enrich_3_t_mat)), .packages = "fgsea", .inorder = TRUE) %dopar% {
fgsea(pathways = geneset_fgsea_hallmark,
stats = enrich_3_t_mat[, i],
minSize = 10,
maxSize = 500)}
stopCluster(cl)BIOSでマルチスレッド(SMT; Simultaneous MultiThreading)をオフにする
上記のRenvironに並列化に関する変数を追加しておけば、おそらくssGESA2もそれにしたがって流れるだろう、とか思っていたのだが、世の中そんなに単純では無いらしい。所詮、自分の知識なんてChatGPTに頼り切ったものってことである。
ssGSEA2など、すでにRパッケージでインストールされているようなものに関しては、うまく行かないらしい。ちなみに、ソースをダウンロードして、doParallelのところに自分でclusterEvalQ()から各変数を記載して、それをソースからインストールしても駄目だった。
# 結局、配布さえているソースにclusterEvalQ()を追加して、そのソースを以下によりインストールしても、ssGSEA2はクラッシュした。
remotes::install_local("/home/kats/ssGSEA2", upgrade = "never", dependencies = TRUE)しかし、シングルスレッドだとssGSEA2がクラッシュしない。「もうこれで良いか….」とか思ったけど、上述の通り「こんなもんに2週間もかけたくないわ!!!」と我に返り、色々と考えることにした。
シングルコアで流れる、ということは、もしかしたらマルチスレッドの設定をなんとかしたらいいんじゃあないか、ということで、ChatGPTに聞いてみたら、BIOSレベルでSMTを切れ、という回答があった。もうわからんので、これに従ってみる。ただし、BIOSの説明書を見なければどこに何があるのか良くわからなったので、説明書をダウンロードし、どの項目が何なのかを色々と調べてみて、ようやくSMT controlを「Disable」にすれば良いことがわかる。ここでマザーボードはASUS X870-P CSM WIFIを使っている。BIOSで以下を設定した。
- Advanced -> AMD CBS -> SMT control -> Disable(これでマルチスレッディングがオフになる。)
- Advanced -> AMD CBS -> Global C-states Comtrol -> Disable(CPUの省電力機能をオフにして、安定的に動作するようにする。)
- Ai tweak -> Precision Boost Override -> Precision Boost Override -> Enable(自動のオーバークロック。最悪、ここもDisableにしなければならない気がする。今回は折角なのでここはEnableにする)
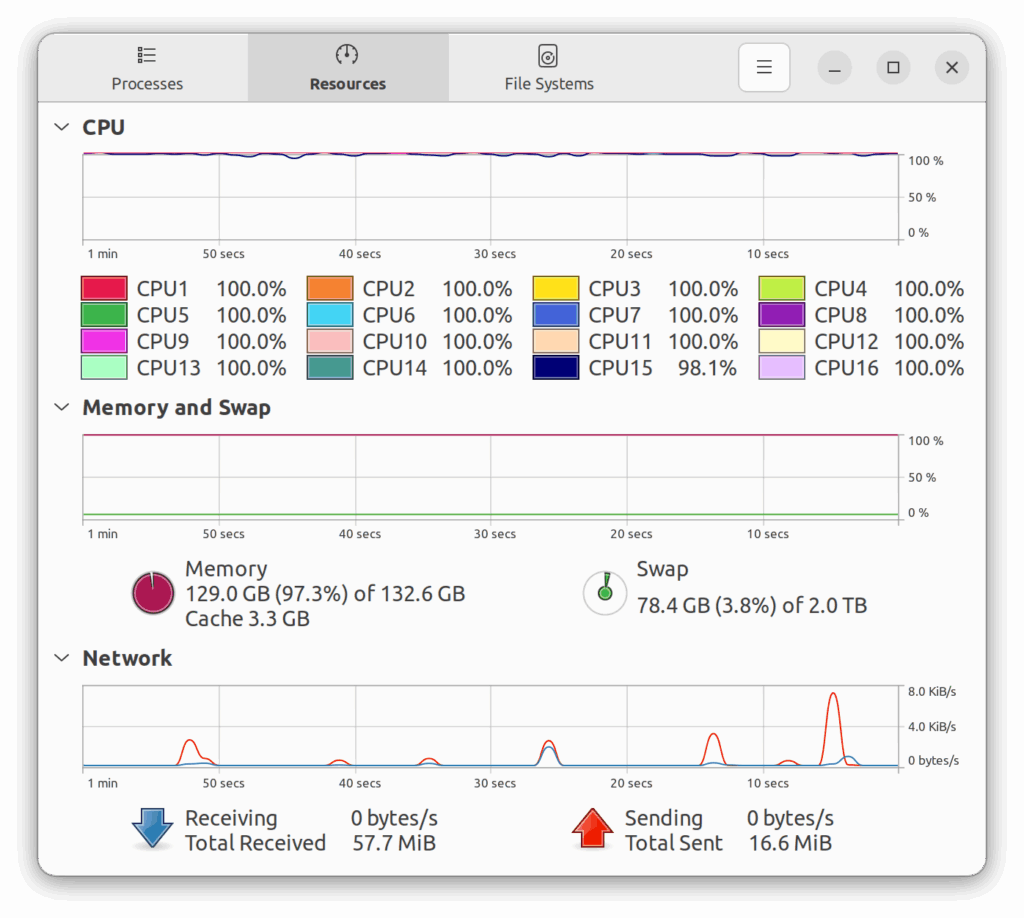
これにより、並列計算を安定化させることが出来るようだ。これによりfgseaもssGSEA2も、ちゃんと並列計算出来るようになった。ひとまず良かった。
他の原因として、メモリ不足なども考えられるが、それはおそらく大丈夫だと思う。foreachは、いちいち全データセットをコピーしてメモリに格納し、そのそれぞれで計算するらしい。すなわち、いちいち5Gバイトくらいあるデータセットを、少なくとも32個格納して計算していくっぽい。ということは、5Gバイトx32スレッド=160Gバイトくらいメモリは必要になる。しかし、このコンピューターはSWAPに2TB設定しているし、そもそも、どうやら並列計算が始まったところで落ちているようだ。なので今回はメモリへの格納に問題はないように思う。
計算速度はどれだけ違うのか
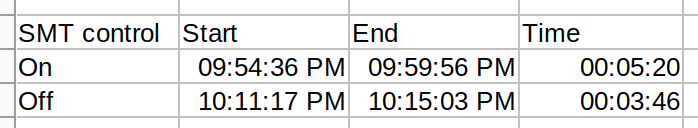
折角の16コア32スレッドなのに…ということで、一応fgseaで速度の比較をしてみた。やったことは単純で、fgseaの実行前後にSys.time()で時刻を取得しただけである。CPU時間をカウントするのも良いけど、実際は何時間費やすか、が知りたいので、こっちでいいように思う。
その結果、どうやらSMT controlを切った方が計算が早い、ということが示された。良かったというか何と言うか。ただし、なんか腑に落ちないんだけど。
2025/11/08(土)並列化が元から組み込まれているライブラリに対してforeachを使うのはそもそも良くない
色々と試行錯誤してきて最終的に至った結論としては、この章代にある通りである。ssGESA2はdoParallel、fgseaはBiocParalelが使用されている。どうやら、このようにそもそも並列化が図られているものに対してforeachを行うと、うまく行かなくなることに気がついた。これに加えて、TCGAのように症例数が10000を超えるデータセットに対して、MsigDBのように35000を超える遺伝子セットを当てようとすると、どうやってもオーバーサブスクリプションのようなことになってしまうようだ(並列計算をしようとするとソフトが落ちる。たまにコンピューター自体も落ちる。メモリもちゃんと動いているらしい。)。もちろん、データセットを分割して並列化処理するという方法もあるようだが、それで計算したところで、予想した計算が出来ているのかを検証する必要も出てきそうなので、こういう場合はおとなしくfor()に切り替えて計算をしたほうが良い。fgseaやssGESA2の並列化を信じて計算が終了するのを待つのが良い。fgseaならnproc=コア数にして、foreachではなくforを使ったコードに直せばよい。
とはいえ、どんな計算でもそうだが、これらに1ヶ月も費やすようならば、それは計算するべきではない。その作業にコンピューターが専有されてしまうし、計算時間が長過ぎる、というのも、昨今ではそのライブラリを使用しない・実用的ではない、と結論するに十分な理由である。大体、職場や大学で、計算に35日かかります。それでも良いですか??とか上司や厳しい教授に言ったら、そんなもの、許可を得ることなんが出来ないし、2週間経ったところでエラーで止まったら、その間が無駄になるわけだ。そんな計算やライブラリなんか、実用的じゃなくて使えんと結論するのが賢いと思う。
まとめ
これでPCやRstudioが突然落ちることなくマルチコアで計算できるようになったのだが、なんか釈然としない。すっきりしない。Ryzen 9 9950Xは16コア32スレッドなのに、16コア16スレッドになる。これを見たとき、なんか悲しくなってきた。でも、こんな計算に1ヶ月費やすよりは良いいか。
おそらく、開発元がインテルのCPUしか使っていなかった場合や、ユーザー数が少ないためにアップデートが重ねられていない場合などにこういう問題って発生するんだろうなって気がする。