- 2025年3月16日
- 2025年3月16日
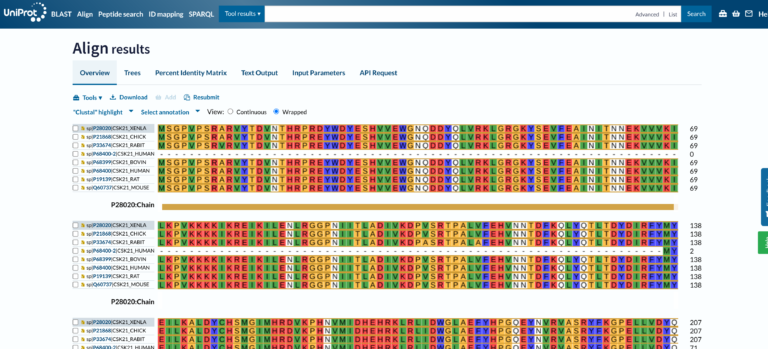
Uniprotでタンパク質のMSA(Multiple Sequence Alignment)を行う
日付;2025/03/16(日) Uniprotを使った簡単なMSA(Multiple Sequence Alighment) 仕事で現在行っているプロジェクトが一段落しそうな雰囲気が出来てきた。この仕事をやりきった上で今年の7月に夏休みをとろうって感じになっている。そして、そのプロジェクトが一段落する時点で、タンパク質-タンパク質相互作用(Protein-Protein Interaction; […]