- 2025年12月14日
- 2026年1月12日
TCGAのデータとRを使ってがんや腫瘍の遺伝子発現量と患者の生存率の関係を解析する(TCGA, v.43, 2025)
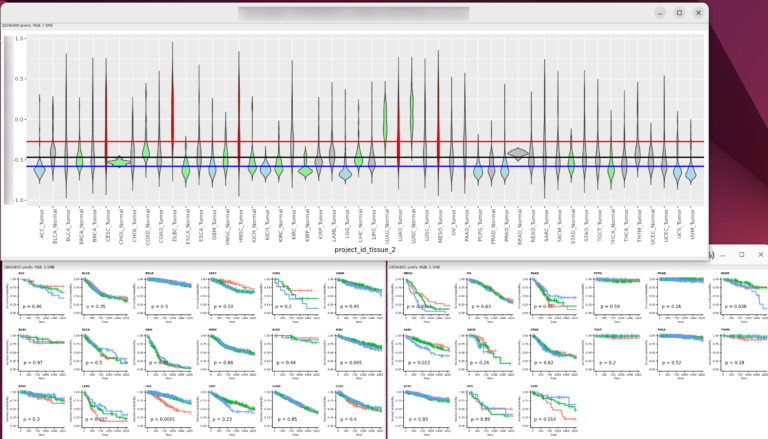
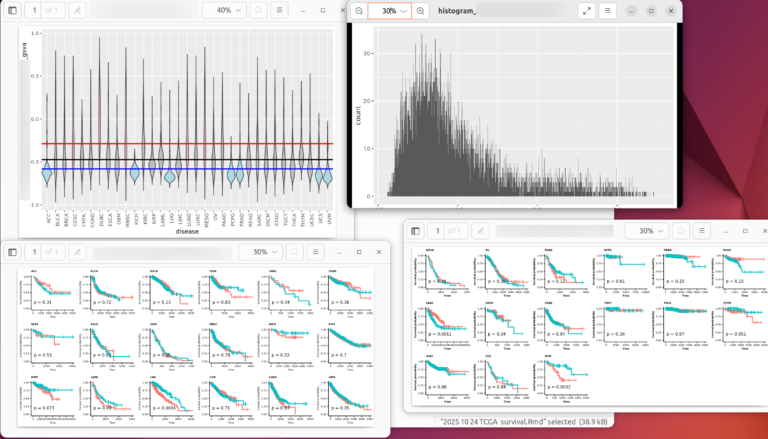
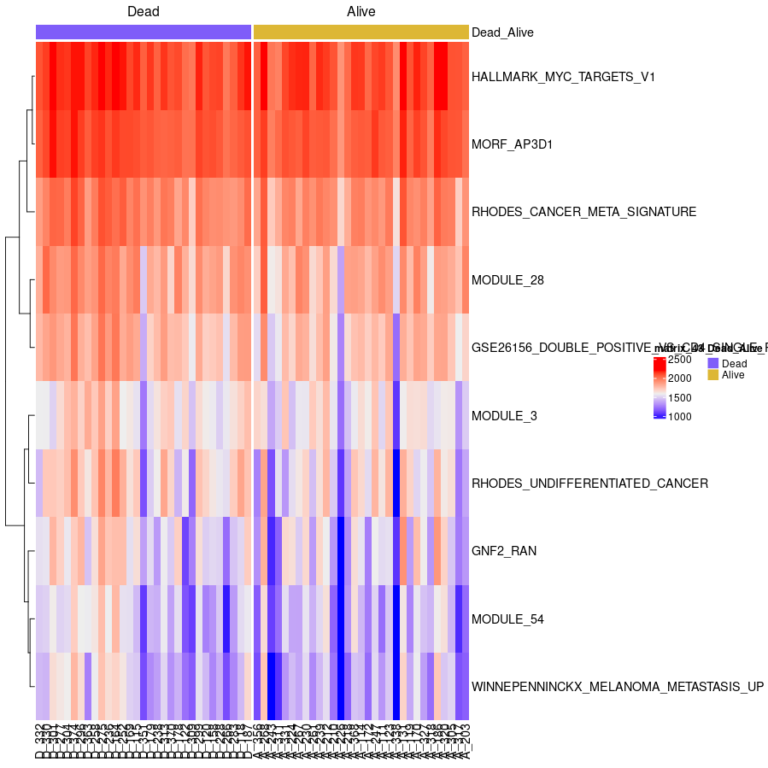
2026/01/03(土);初稿2026/01/12(月);追記;AnndataではなくSummerizeExperimentやMultipleAssayExperimentが使えると思う。 はじめに 以前、TCGAのデータを使って、がんや腫瘍における遺伝子発現量(RNA-seqのカウント値)と患者の生存率の関係をRで解析したが、そのときは対応する正常組織(おそらくがんや腫瘍周辺の正常部位)におけ […]