日付;2022/10/30(日)、2024/03/31(日)有料化
Single cell RNA sequence(scRNA-seq)の解析を行うための代表的なソフトの一つにmonocle3がある。最も王道のツールとしてSeurat、その次にこのmonocle3が挙げられるような感じがする。実は、Seuratもmonocle3も、マニュアルに目を通した程度しか知らないのだが、印象としてはこのmonocle3のほうが扱い易いのではないかと思っている。これら2つとscanpyなんかも試す必要があるように思っている。
そこで、monocle3をUbuntu20.04にインストールしようと思うが、Seuratと同様、というかそれ以上に煩雑だったので記録をしてく。
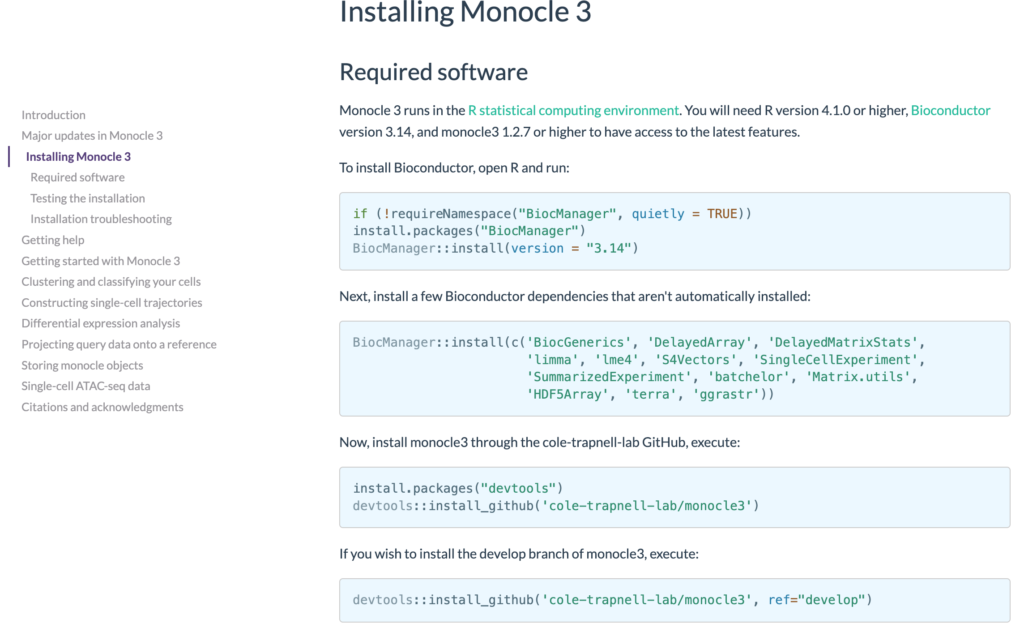
インストールの手順は、monocle3に従う。Bioconductorからいくつかのライブラリーをインストールし、次にGithubからmonocle3をインストールすれば良い。インストール方法は、Seuratをインストールしたときと同じである。必要ならばここを参照にすると良い。簡単に述べると、基本Rstudioを使わず、ターミナルでsudo apt searchとsudo apt installを使ってインストールしていく。そしてRが必要ならがターミナルでsudo Rを使い、それでも、例えば、必要なライブラリやパッケージが揃っているにも関わらず依存関係のエラーが出る場合はRstudioでインストールする。この方法でインストールされたRのパッケージは少し古いものになるが、このような複雑かつ面倒なパッケージをUbuntuに入れるときにはこれで良い。