日付;2021/05/31(月)
昨日、Macbook Pro 15 inch (Mid 2015) (MBP15)にDebian 9をインストールし、RNA-seqのプリプロセッシングに必要なソフトをインストールした。方法は過去にVirtualBox中のDebain 9にインストールしたときとほぼ同じであった。
このMBP15は、CPU intel core i7-2620M (2 cores 4 threads)、メモリ16GBだ。STARでRNA-seqをマッピングするためにはどのくらい時間が必要なのか目安を知ろうと思う。
STARのバージョンは2.7.7a。これは以前M4800に入っていたSTARと同じバージョンである。今考えてみれば、もう新しいヤツでもよかったかなぁと思っている。
リファレンスのゲノムとしてはSTARのマニュアル通り、GENECODEのPrimary assemblyを使った。ヒト、マウスのゲノムは、それぞれRelease 38 (GRCh38.p13)、Release M27 (GRCm39)を使用した。アノテーションファイルとしてGTFを用い、ヒトではこれ、マウスはこれ、リファレンスのゲノムはヒトではこれ、マウスではこれ。これらは現時点(2021年5月31日)で最新のリファレンスである。
まず前置きとしてSTARを使うのにメモリ16GBは無謀である。CPUについても調べてみてこんなに遅いとは思わなかった。
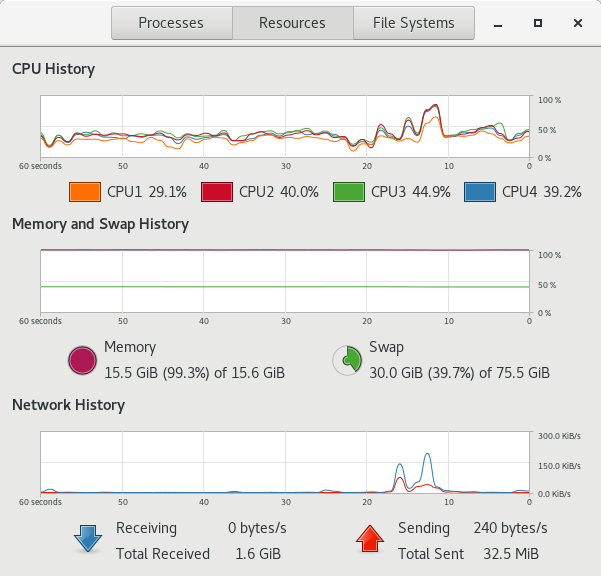
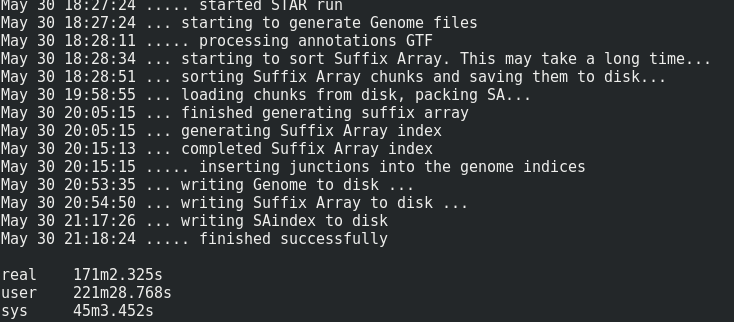
マウスのゲノムインデックス作成の結果が以下。
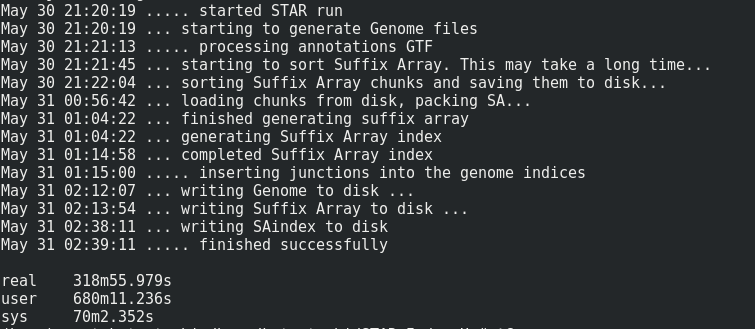
ヒトの場合は以下。
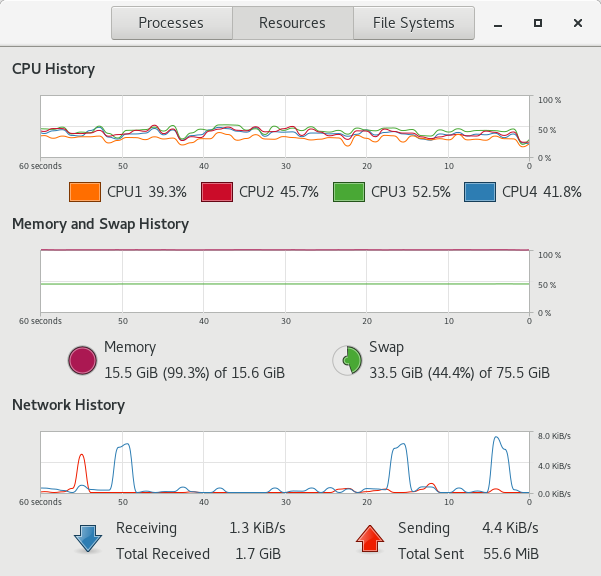
この記事を書いていて気がついた。STARの設定、間違えた。8 threadsで計算するように設定してしまった。もしかしたら、こんなに遅いのはこれが原因の一つかもしれない。なんにしても、かなりかかる。これはまずい…
マニュアルには物理メモリは最低32GB必要と書いてあるが、それでは足りない。物理メモリのみで計算する場合は64GB必要だとわかった。しかし、今の時代、ストレージはSSDとかになるだろうから、R以外を使うぶんにはSWAP領域で対応できるかもしれない。一方、CPUの計算速度がどうにもならん。おそらくマッピングは、マウスの場合はなんとかなりそうだが、ヒトの場合は3、4時間かかってしまうかもしれない。これが合計10ファイルあったら40時間必要ってことになる。怖いなぁ〜。2コア4スレッドて、しかし…今度はマッピングについてメモリの使用量の時間経過もとってみよう。というか、なんでとらなかったんだろう。
何はともあれ、なんとかなることがわかった。自分はbioinformaticianではないし、こういった解析に関しては多くのbiologistがやっているようにエクセルで平均値と標準誤差をバカのひとつ覚えのように計算するような行為の延長と考えいてる。なので、遅かろうが一向にかまわん。もし何か言われたら、「おそらくマッピングで計算が4日、3次解析が7日、週末込で2週間くらいかかると思います。」と答えようと思う。そうじゃなかったら、自分ではなく、低クオリティーなTechnitianに頼んでください。