2024/10/27(日);初稿
2026/01/06(火);データのバージョン追記、タイトルを変更、サムネイル変更
はじめに
最近では、ナノポアシークエンサーでアミノ酸配列まで読めるようになってきた。これはつまり、プロテオームもハイスループットで読める時代がもうそこまで来てるってことである。現時点ではマルチプレックスのプロテオームはRNAシークエンスの価格の10倍くらいするので、なかなか手を出せる物でもない。しかし、こうやってハイスループットのタンパク質発現解析方法が開発されれば、数年後にはサンプルあたりのリードの価格も随分下がり、これまではTCGAのような遺伝子解析のデータベースで行われていたトランスレーションが、タンパク質発現で行えるようになる。
このCPTAC(Clinical Proteomic Tumor Analysis Consortium)は、各疾患のタンパク質発現や修飾に関して集めた、TCGAと同じようにして扱うことができる公開データである。現時点では遺伝子発現や変異の解析が価格や網羅性などで優位であることもあり、TCGAや他のデータベースと似ているプロテオームの解析に目が行くように思うが、その他にもユビキチンやメタボロームの項目があり、今後の発展にすごく期待が持てるデータベースである。遺伝子数や症例数はTCGAのほうが勝っているが、そうは言っても既に十分に解析に使用できるまでになっている。
別の記事でTCGAのデータのダウンロード方法について書いたが、ここではCPTACのダウンロード方法を記録しておく。
CPTAC
CPTACはここ(https://proteomics.cancer.gov/programs/cptac)にある。ここの右側中央にある「Data Portal」というところから、PDC(Proteomics Data Commons)ポータルに行く。ちなみに、TCGAなどがGenomics Data Portalというところである。
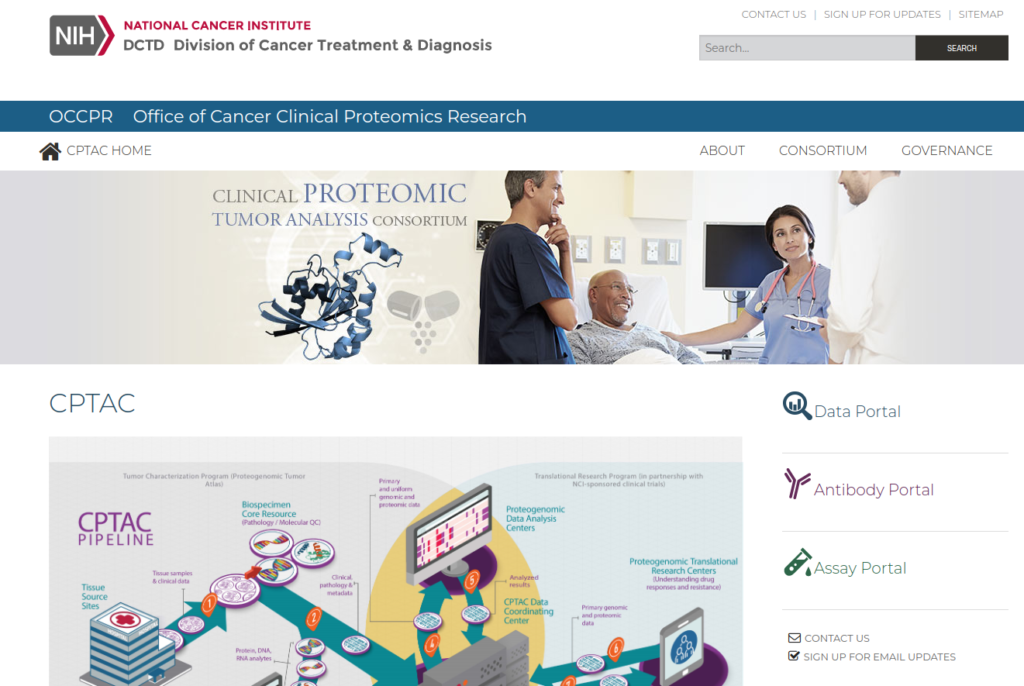
Data Portalに何か色々と書いてある。その下にある「PDC」を押す。
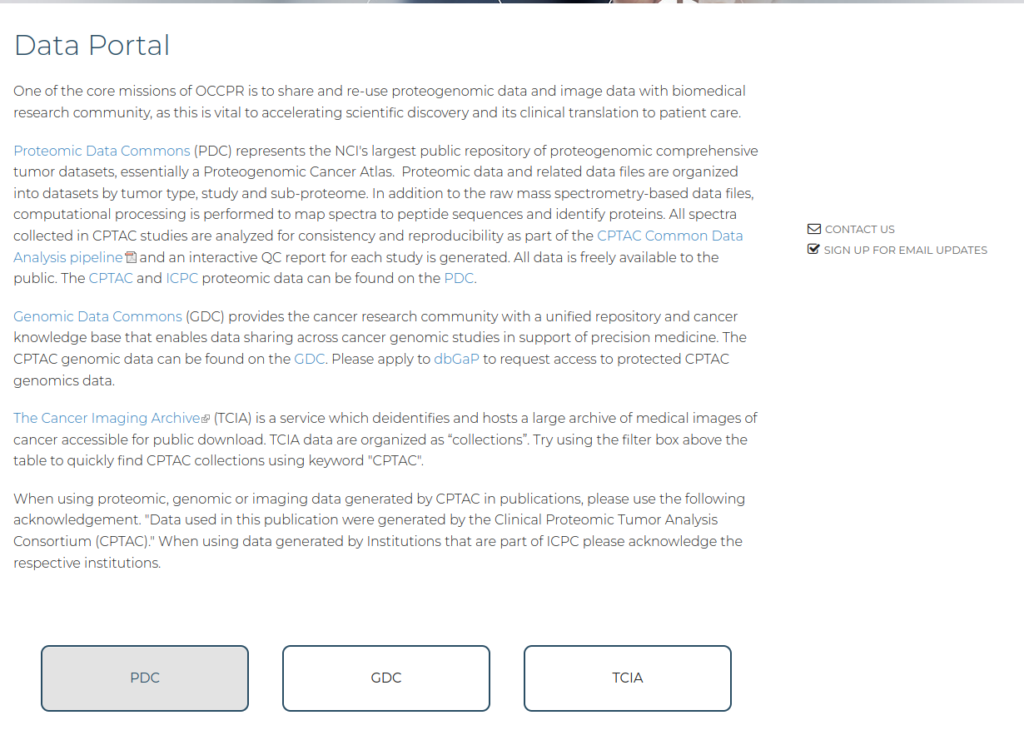
次に、「EXPLORE」を押す。
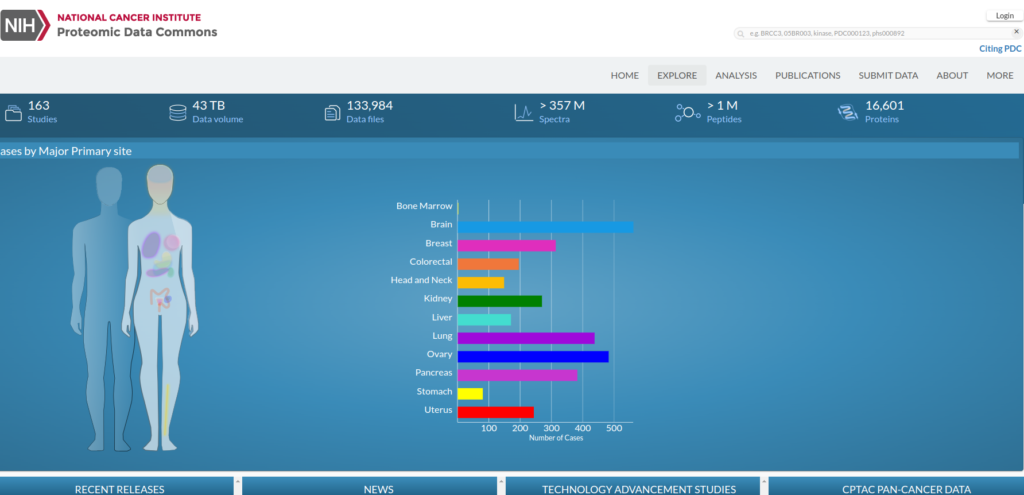
EXPLOREを押すと、次に以下に来る。ここで目的の症例を選ぶことになる。
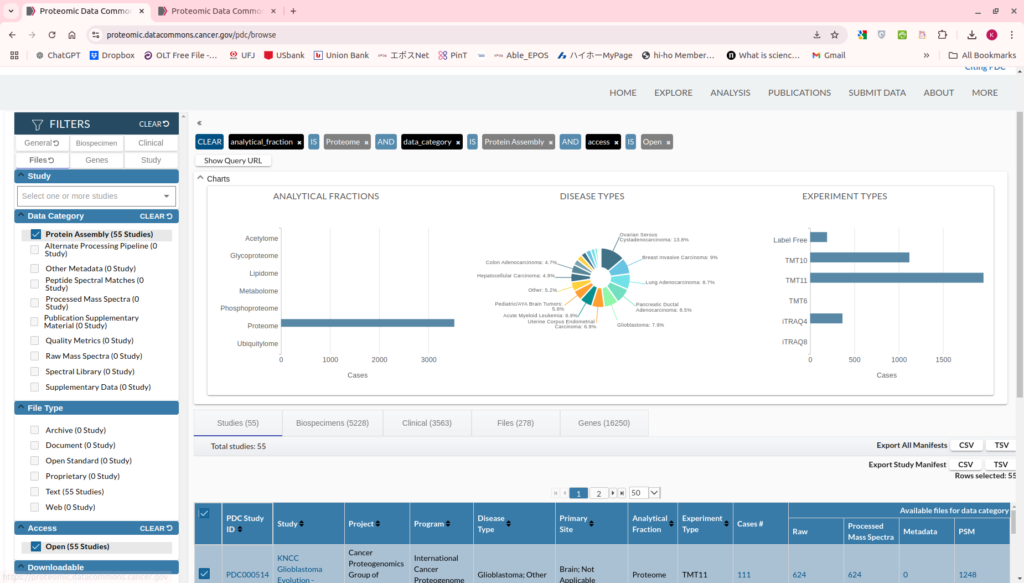
今回は以下の項目を選ぶことにする。
| FILTERS | 項目1 | 項目2 |
|---|---|---|
| General | Analytical Fraction | Proteome |
| Files | Data Category | Protein Assenbly |
| Files | Access | Open |
ここで、個人的に抑えておかなくてはならないのが、FilterのGeneのところである。Geneのところには、例えば、TP53という遺伝子を入力すると、どんな遺伝子ID、例えばNP_000537やENSP00000478499がTP53であるか、などがまとめられたテーブルが入手できる。ただ、それだけである。最も必要なのはTP53のような遺伝子名(タンパク質名)だけであるように思う。
次に、Study、Biospeciemen、Clinical、Files、Genesのタブを押し、その下の青でハイライトされたテーブルの左上にチェックを入れて、すべてのレコードを選択しExport File ManifestにあるTCVをダウンロードする。欲張って全部ダウンロードしようとすると、ファイルが多すぎるというエラーが出る。
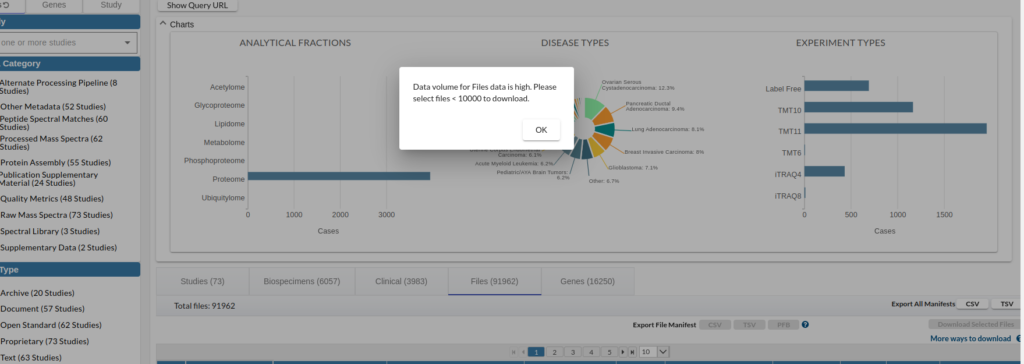
欲張って全部入手しようとしても、このようなエラーがでる。
これで、無事にManifestファイルを入手できる。
pdc-clientのインストールとデータのダウンロード
gdc-clientのときと同様、Windows(windows 10), Mac (MacOS Monterey), Ubuntu 22.04について述べる。各々のOSでダウンロードの方法を記すが、自分はメインでUbuntu 22.04を使っているので、その使い方を中心に述べる。
pdc-clientはここ(https://proteomic.datacommons.cancer.gov/pdc/data-download-documentation)からダウンロードできる。注意点としてはpdc-clientだけでなくUI(User Interface)も一緒にダウンロードされてくるので、CL(client)の方を使えば良いと思う。わざわざ重いUIを使ってストレスを感じる必要はない。
また、ここ(https://proteomic.datacommons.cancer.gov/pdc/faq/Data_Download)には、各ファイルは一体何が書いてあるので、解析する際に読めば良いと思う。
Windows (Windows 10)
インストール
上記のURLから圧縮されたバイナリをダウンロードし、任意のディレクトリ(フォルダ)へ展開する。
そして、そのディレクトリのなかにManifestファイルも入れておく。PDC000121とか書いてあるフォルダが、pdc-clientによりダウンロードされてきたデータである。
使い方
pdc-clientが保存されているディレクトリにchdirで移動し、pdc-clientを走らせる。TCGAのときにせっかく環境変数からパスを通しているのに全然機能しなかったので、今回はそれはやらなかった。
chdir C:\Users\Someone\PDC
pdc-client.exe download -m PDC_file_manifest_10062024_003500.tsv --log-file LOG_FILEMac (MacOS Monterey)
これもTCGAのダウンロードで使ったgdc-clientと同じである。バイナリをダウンロードして展開し、それを任意のディレクトリに保存しておく。そして、保存しているディレクトリに行って以下の画像にあるコマンドで実行する。ところが、実行すると以下のようなメッセージが返ってきた。
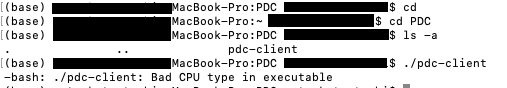
まず、System Requirementを見てみると以下2024年10月27日(日)の時点では以下のように書いてある。これを見てどう思うかはユーザー次第なんだろうけど、自分は、古いと思う。そして、自分の持っているMacは、MacOS X Monetery(12.7.9)である。
- OS: Linux (Ubuntu 14.x or later, CentOS 7), OS X (10.9 Mavericks or < 12.0 ), or Windows (7 or later)
- Please delete any previous instances of the download client before downloading a newer version.
こういうことがあるから、この手の解析はLinux(UbuntuもしくはDebian。Ubuntuの方が楽だけど、Debianの方が面白い。)一択である。Bad CPU type in executableって、どっちかって言えば悪いのはそっちだろう。対応OS古くないか?Apple M3とか使っているヒトはどうしてるんだろう。
ここでの結論は「現時点(2024年10月27日)ではpdc-clientはMacでは使えない」である。NCIさんは早いところpdc-clientのアップグレードしたほうが良いのではないかと思う。
Ubuntu (Ubuntu 22.04)
インストール
バイナリをダウンロード、展開、pdc-clientを任意のディレクトリに保存する。次に以下でパスを通し、有効化する。それでOK。考えるのが面倒なので、自分はhome直下に入れた。
cd
sudo gedit .bashrc
# Add PATH=$PATH:/home/kats/pdc-client into .bashrc, and then save.
source .bashrc # to be effective the path
#check path using follow.
echo $PATH使い方
これはTCGAのgdc-clientと全く同じ。
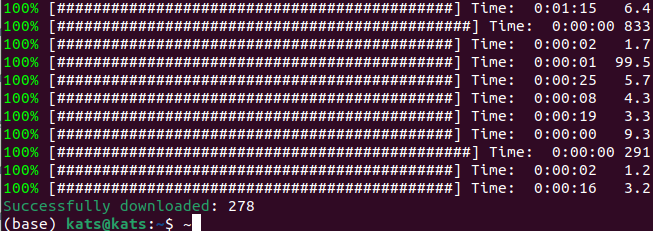
pdc-client download -m "/mnt/Blog/CPTAC/PDC_file_manifest_10202024_102427.tsv" -d "/mnt/Blog/CPTAC/count" --log-file "/mnt/Blog/CPTAC/PDC_file_manifest_10202024_102427_log.txt"
上手くダウンロードできたら、Successfully downloadedとなる。TCGA(GDCに保存されているデータ)のときは各症例毎にダウンロードされて来てかなり大量のダウンロードになったが、CPTAC(PDCに保存されているデータ)はコホートごとのダウンロードになるので、ファイル数が少ない。ただし、各々のファイルサイズはその分大きい。
同じ症例の遺伝子発現データがGDC portalからダウンロードできる。
医学生物学研究を行っていると絶対に聞かれる以下のような質問がある。
「これって遺伝子発現の結果であって、タンパク質発現じゃないですよね。機能に言及するならタンパク質発現を見ないと意味ないですよね?」
教授だとしても、これを言ってくる奴は勉強不足である。はっきり言うが、もう駄目である。研究者としての大脳新皮質、特に前頭前野あたりの萎縮もしくは腐敗が進んでいる証拠であると思う。どの分野にも絶対居る。なんやったら部屋毎に一人は居る。まず、この発言をする人間は目先しか考えておらず、いろいろな可能性を捨てている。それに、これを言う人間こそが過去に自分が言われたことを何も考えずに口に出している、バイアスで動いている人間であると思う。「基本的にタンパク質は遺伝子の転写がもとなんだから、とりあえずは相関するはずである。」と順々に考え、その次に「いうても修飾があるし」となり、でも「細胞表面マーカーとかだと相関しない場合もあるけどな」などといった経験と照らし合わせると考えたほうが柔軟で良い気がする。
言うても、そういうことが心配(絶対に誰かがコメントしたから解析しているような状態だとは思うが)ならばGDC portalに行って、TCGAの代わりにCPTACを選んでそのRNA-seqの結果をダウンロードしてきて、疾患のマッチングをして関心のあるタンパク質と遺伝子の発現の相関を解析して示してやれば良い。CPTAC-2とCPTAC-3があるが、CPTAC−3では遺伝子発現も取得されている症例も多く、実際にケースマッチングも相関の解析も可能である。
予想出来る質問ならば、準備しておいて叩きのめしてやるのは結構好きである。最終的に印象が悪くなるとか、どうでも良い。
ちなみに、上記に質問をしてくる奴って、タンパク質ではなくその遺伝子発現が思った挙動をしていない場合でも「なぜこの遺伝子発現が変動していないんだ?」とか言ってくる。お前、この間遺伝子発現だから関係ねえって言ってたじゃあねえのかよ。こういうところが、前頭前野腐敗バイアス野郎っていうんだよ。感じるな。考えろ。
2026/01/06(火)追記;データのバージョンについて
ここでダウンロードしたのはV4.4 (October 3, 2024)のCPTACのデータである。ここ(https://pdc-release-notes.s3.amazonaws.com/PDC_Data_Release_Notes.htm)に各データのリリースノートが記載されている。論文や報告書には絶対にバージョンは書かなくてはならないので、バージョンは記録しておく必要が有る。
まとめ
TCGAは既にトランスレーションのためにはならないデータセットになっている。そしてこのCPTACはTCGAよりも更に強力なツールになるに決まっている。さらに、GDCとPDCにあるデータの両方を使うことができればそれは最強である。そしてこれを知っている者と知らない者では、その差は歴然だろう。