日付;2022/08/28(日)
次はUbuntu 20.04.4に、よく使うRNA-seq解析用のソフトをインストールしていく。何か追加でインストールした場合は、ここに追加で記録していくことにする。
RNA-seqのプリプロセッシング用ソフトウェア一式
基本的に、WSL2でUbuntuを入れ、そこにRNA-seqで使用するソフトウェアを入れたときとほぼ同じようにインストールできる(ここ)。まずインストールしたソフトは以下である。fusioncatcher以外はなんの問題もなくインストールできた。
fastqc
trimmomatic
prinseq++
star
subread
samtools
fusioncatcher
ngs_disambidugate
bwa
bowtie2
bowtie
fastq screen
rseqc
fusionCatcher
しかし、fusionCatcherでつまずいた。エラーを見ていると、どうやらBiopythonのインストールが上手くいっていない気がする。なので、以前Ubuntu@WSL2で使った方法に加えて、Biopythonもインストールした。そうしたら、上手くインストールできた。マニュアルを見ている限り、頭のapt installも、まぁ、何が必要なのかわからんので、とりあえず流してみる。ただし、どうやらもうUbuntuではなくなっているパッケージもあるみたいで、上手いことインストールができなかった。個人的に、このfusionCatcherというヤツはどんどん古くなってきているように思う。今は動くから良いけど、数年後はわからん。代価の良いソフトを見つけたほうが良いかもしれない。
sudo apt install wget gawk gcc g++ make cmake automake curl unzip zip bzip2 tar gzip pigz parallel build-essential libncurses5-dev libc6-dev zlib1g zlib1g-dev libtbb-dev libtbb2 python python-dev python-numpy python-biopython python-xlrd python-openpyxl default-jdk ##python-xlrdあたりでエラーがでる。
conda activate py27
wget http://sf.net/projects/fusioncatcher/files/bootstrap.py -O bootstrap.py && python bootstrap.py -t --download
cd /home/katsu/fusioncatcher/test
conda install -c conda-forge biopython #Ubuntu@wsl2ではこれは必要なかったが、Ubuntu 20.04.4のデスクトップバージョンでは必要らしい。
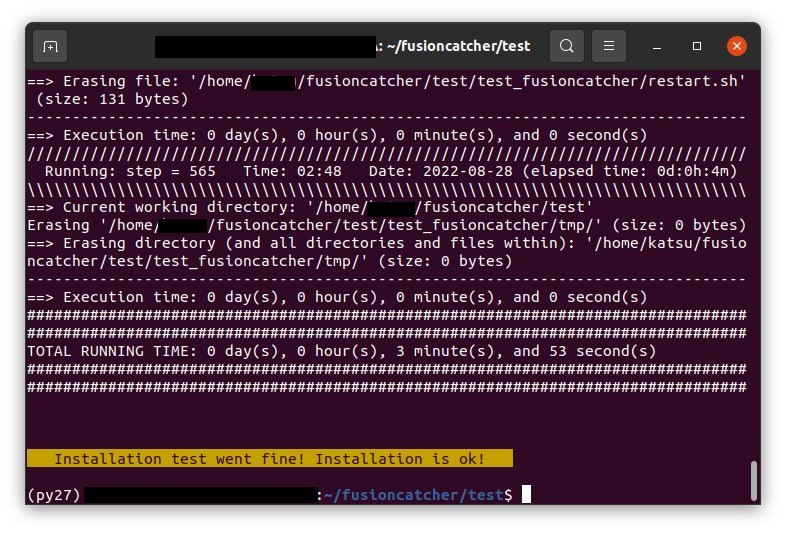
./test.sh
conda deactivate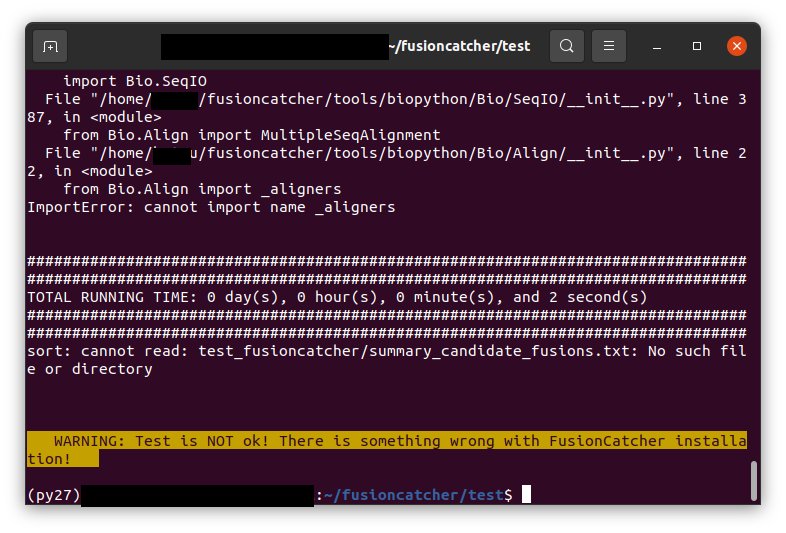
Subread
Subreadを入れえるのを忘れていたので、それも入れた。バイナリーなので、展開後にPATHを通せば良い。これはfeaturecountsで必要。
wget https://sourceforge.net/projects/subread/files/subread-2.0.3/subread-2.0.3-Linux-x86_64.tar.gz
tar zxvf subread-1.x.x.tar.gzmultiqc
これもけっこう便利。QCの結果をまとめてくれるので、誰かに説明するにはすごく便利である。
conda install -c bioconda -c conda-forge multiq